Ob Pollenflugvorhersage, Honiganalyse oder das Verstehen klimabedingter Veränderungen von Pflanzen-Bestäuber-Interaktionen - die Analyse von Blütenpollen spielt in vielen Forschungsbereichen eine wichtige Rolle. Goldstandard ist dabei nach wie vor die Mikroskopie, die jedoch viel Zeit und Expertise erfordert.
Wissenschaftlerinnen und Wissenschaftler des Helmholtz-Zentrums für Umweltforschung (UFZ) und des Deutschen Zentrums für integrative Biodiversitätsforschung (iDiv) haben in Kooperation mit der Technischen Universität (TU) Ilmenau nun ein Verfahren entwickelt, mit dem sie die Pollenanalyse automatisieren können. Ihre Studie ist im Fachmagazin New Phytologist veröffentlicht.
Pollen wird in den Staubblättern einer Blüte produziert und besteht aus vielen winzigen Pollenkörnern, die das männliche Erbgut einer Pflanze für deren Fortpflanzung beherbergen. Die Pollenkörner verfangen sich in den Härchen vorbeistreifender nektarsaugender Insekten und werden so von Blüte zu Blüte transportiert. Dort bleibt er im besten Fall an der klebrigen Narbe derselben Pflanzenart hängen, und es kann zu einer Befruchtung kommen.
„Die bestäubenden Insekten machen diesen Pollen-Kurierdienst zwar ganz nebenbei, doch ist er von unschätzbar hohem ökologischen und auch wirtschaftlichen Wert“, sagt Dr. Susanne Dunker, Leiterin der Arbeitsgruppe Bildbasierte Zytometrie im Department Physiologische Diversität an UFZ und iDiv. „Insbesondere vor dem Hintergrund des Klimawandels und dem zunehmenden Verlust von Arten ist es wichtig, diese Pflanzen-Bestäuber-Interaktionen besser zu verstehen.“ Die Pollenanalyse ist dafür ein entscheidendes Instrument.
Pollenkörner haben eine für die jeweilige Pflanzenart charakteristische Form, Oberflächenstruktur und Größe. Um die zwischen 10 und 180 Mikrometer großen Pollenkörner einer Probe zu bestimmen und zu zählen, galt bislang die Mikroskopie als Goldstandard. Die Arbeit am Mikroskop erfordert jedoch eine große Expertise und ist sehr zeitaufwendig.
„Zwar gibt es bereits verschiedene Ansätze zur Automatisierung der Pollenanalyse, doch können diese Methoden entweder nah verwandte Arten nicht unterscheiden oder keine quantitativen Aussagen über die Anzahl der in einer Probe enthaltenen Pollenkörner treffen“, sagt die UFZ-Biologin Dunker. Doch genau das ist für viele Fragestellungen in der Forschung, etwa zur Interaktion zwischen Pflanzen und Bestäubern, wichtig.
In ihrer aktuellen Studie hat das Forscherteam um Susanne Dunker ein neuartiges Automatisierungsverfahren für die Pollenanalyse entwickelt. Dafür kombinierte es den Hochdurchsatz der bildbasierten Durchflusszytometrie, ein Verfahren der Partikelanalyse, mit einer Form der künstlichen Intelligenz (KI), dem sogenannten Deep Learning – und entwarf damit ein hocheffizientes Analyse-Tool, das neben der exakten Artbestimmung auch eine Quantifizierung der in einer Probe enthaltenen Pollenkörner möglich macht.
Die bildbasierte Durchflusszytometrie ist ein vorwiegend in der Medizin angewandtes Verfahren zur Analyse von Blutzellen, das nun auch der Pollenanalyse dient. „Eine zu untersuchende Pollenprobe wird zunächst in eine Trägerflüssigkeit gegeben, die dann einen enger werdenden Kanal durchfließt“, erklärt Susanne Dunker dieses Verfahren.
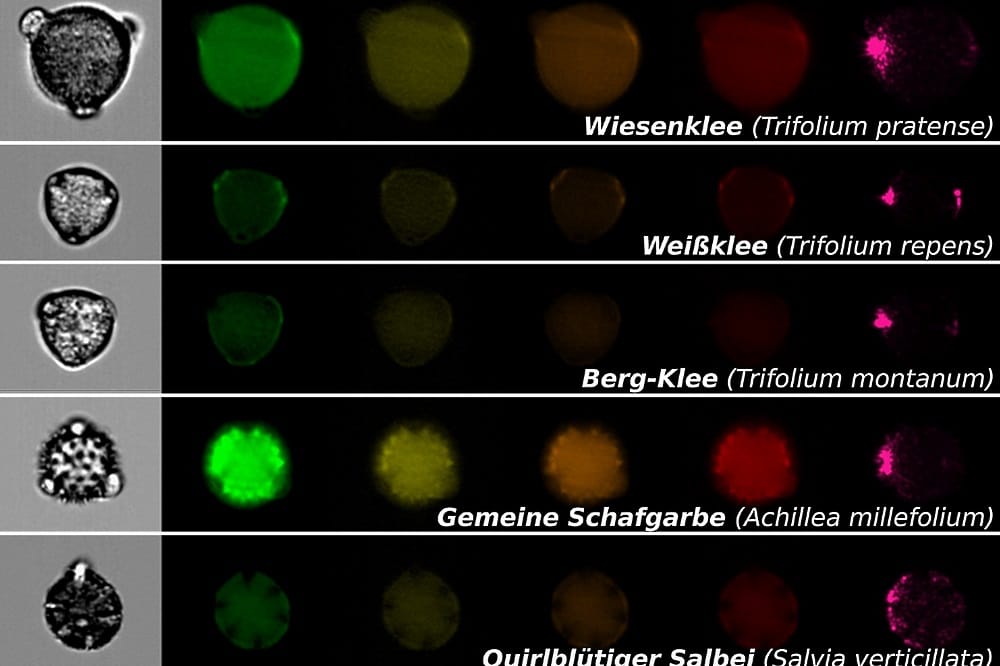
„Durch die Verengung, werden die Pollenkörner separiert und wie auf einer Perlenschnur aufgereiht. So wandert jedes Pollenkorn einzeln durch das eingebaute Mikroskop-Element – bis zu 2.000 Pollen pro Sekunde können das sein.“ Zwei normale mikroskopische Bilder werden mit zehn fluoreszenzmikroskopischen Aufnahmen pro Pollen ergänzt. Dabei senden die Pollen, nachdem sie durch Laser mit Licht bestimmter Wellenlängen angeregt wurden, selbst Licht aus.
„In welchem Bereich des Farbspektrums und wo genau die Pollen fluoreszieren, ist teilweise sehr spezifisch. Damit erhalten wir weitere Merkmale, die zur Identifizierung der jeweiligen Pflanzenart beitragen können“, sagt Susanne Dunker. Beim Deep learning abstrahiert ein Algorithmus in aufeinanderfolgenden Schritten die ursprünglichen Pixel eines Bildes immer stärker, um letztendlich die für eine Art spezifischen Eigenschaften zu extrahieren.
„Die kombinierte Nutzung von mikroskopischen Aufnahmen, Fluoreszenzeigenschaften und Hochdurchsatz gab es in der Pollenanalyse bislang noch nicht – das ist tatsächlich ein absolutes Novum.“ Die Analyse einer wenig komplexen Probe dauert beispielsweise vier Stunden am Mikroskop, mit dem neuen Verfahren sind es nur noch 20 Minuten. Das neuartige Hochdurchsatz-Analyseverfahren ist deshalb als Patent des UFZ angemeldet, Susanne Dunker erhielt 2019 dafür den Technologietransfer-Preis des UFZ.
Die in der New Phytologist-Studie untersuchten Pollenproben stammten von 35 Wiesenpflanzenarten, darunter zum Beispiel Schafgarbe, Salbei, Thymian und verschiedene Klee-Arten wie Weiß-, Berg- und Wiesenklee. Insgesamt fertigten die Forscher rund 430.000 Bilder an, die den Grundstock für eine Datenbank bildeten. Diese wurde in Kooperation mit der TU Ilmenau mittels Deep Learning in ein hocheffizientes Werkzeug zur Pollenidentifizierung überführt.
In anschließenden Untersuchungen testeten die Forscher die Treffsicherheit ihrer neuen Methode: Aus den 35 Pflanzenarten haben sie unbekannte Pollenproben mit der Datenbank verglichen. „Das Ergebnis war mehr als zufriedenstellend, die Genauigkeit lag bei 96 Prozent“, sagt Susanne Dunker. Dabei konnten sogar Arten, die selbst für Experten am Mikroskop schwer zu unterscheiden sind, sicher identifiziert werden. Die neue Methode ist also nicht nur extrem schnell, sondern auch hochpräzise.
Das neue Verfahren der automatisierten Pollenanalyse wird künftig bei wichtigen Forschungsfragen rund um die Pflanzen-Bestäuber-Interaktionen eine zentrale Rolle spielen. Wie wichtig sind bestimmte Bestäuber wie Biene, Fliege oder Hummel für eine Pflanzenart? Welche Konsequenzen hätte ein Verlust einer bestäubenden Insekten-Art oder einer Pflanze?
„Wir sind nun in der Lage, in großem Umfang Pollenproben qualitativ und gleichzeitig quantitativ auszuwerten. Unsere Pollen-Datenbank insektenbestäubter Pflanzen erweitern wir dafür stetig“, sagt Susanne Dunker. Sie will die Datenbank auf mindestens jene 500 Pflanzenarten erweitern, deren Pollen für die Honigbiene als Nahrung relevant sind.
Publikation:
Susanne Dunker, Elena Motivans, Demetra Rakosy, David Boho, Patrick Mäder, Thomas Hornick, Tiffany M. Knight: Pollen analysis using multispectral imaging flow cytometry and deep learning. New Phytologist https://doi.org






Keine Kommentare bisher