Detaillierte Informationen zur Landbedeckung sind wichtig für ein besseres Verständnis unserer Umwelt - etwa zur Abschätzung von Ökosystemleistungen wie Bestäubung oder um Nitrat- und Nährstoffeinträge in Gewässer zu quantifizieren. Diese Informationen werden zunehmend aus zeitlich und räumlich hochaufgelösten Satellitenbildern gewonnen.
Häufig versperren jedoch Wolken den Blick aus dem All auf die Erdoberfläche. Eine dynamische Anwendung von Methoden des maschinellen Lernens kann das lokale Auftreten von Wolken berücksichtigen, ohne auf bislang genutzte Interpolationsverfahren zurückzugreifen. Das zeigen Wissenschaftler des UFZ in einer Studie, die im Fachblatt Remote Sensing of Environment veröffentlicht wurde. Ihr Algorithmus erkennt 19 verschiedene Feldfruchtarten mit einer Genauigkeit von 88 Prozent.
“Wenn wir feststellen können, was auf den Feldern einer Region wächst, können wir nicht nur auf den Nährstoffbedarf, sondern auch auf die Nitratbelastung umliegender Gewässer schließen”, erklärt Sebastian Preidl, Wissenschaftler im Department Landschaftsökologie am UFZ. Auch ließen sich mit den Informationen beispielsweise konkrete Maßnahmen zum Schutz von Wildbienenbeständen besser initiieren. “Nur wenn wir die biologische Vielfalt einer Region kennen, können wir sie effektiver schützen”, so Preidl.
Erdbeobachtungssatelliten, wie die des Copernicus Programms der europäischen Weltraumorganisation ESA, liefern zeit- und räumlich hochaufgelöste Daten und machen damit ein kontinuierliches Monitoring der Landoberfläche auf ökologisch relevanter Skala möglich. So werden vom Satelliten Sentinel-2, auf dessen Daten sich Preidls Arbeit stützt, regelmäßig Bilder der Landoberfläche in neun verschiedenen Spektralbereichen aufgenommen. Aus solch einer spektralen Zeitserie können die Forscher schließen, womit der Boden im betreffenden Gebiet bedeckt ist.
Eine zentrale Herausforderung im Umgang mit Zeitreihen optischer Satellitendaten ist die Beeinträchtigung durch Wolken. Trotz zahlreicher Satellitenaufnahmen führt häufige Bewölkung dazu, dass größere Datenlücken in den spektralen Zeitreihen auftreten. Gleichwohl ist eine ausreichende Zahl an Bildpixeln (Beobachtungen) zu möglichst vielen Pflanzenwachstumsphasen notwendig, um die aufgenommenen spektralen Signaturen den entsprechenden Pflanzenarten zuzuordnen.
Diese Lücken schließen üblicherweise künstlich erzeugte Daten, die aus wolkenfreien Bildpixelwerten interpoliert werden. “Wir verzichten darauf und haben uns stattdessen für eine dynamische Anwendung von Modellen des maschinellen Lernens entschieden. Das heißt, wir generieren maßgeschneiderte Algorithmen für jedes Pixel”, sagt Preidl.
“Unser Algorithmus sucht sich wolkenfreie Pixel automatisch aus dem gesamten Satellitenbilddatensatz heraus und ist nicht auf großflächig wolkenfreie Szenen angewiesen. Um jedem Bildpixel eine spezielle Feldfrucht zuzuweisen, wird die zeitliche Abfolge von wolkenfreien Beobachtungen auf Pixelebene von einer Vielzahl von Modellen berücksichtigt.”
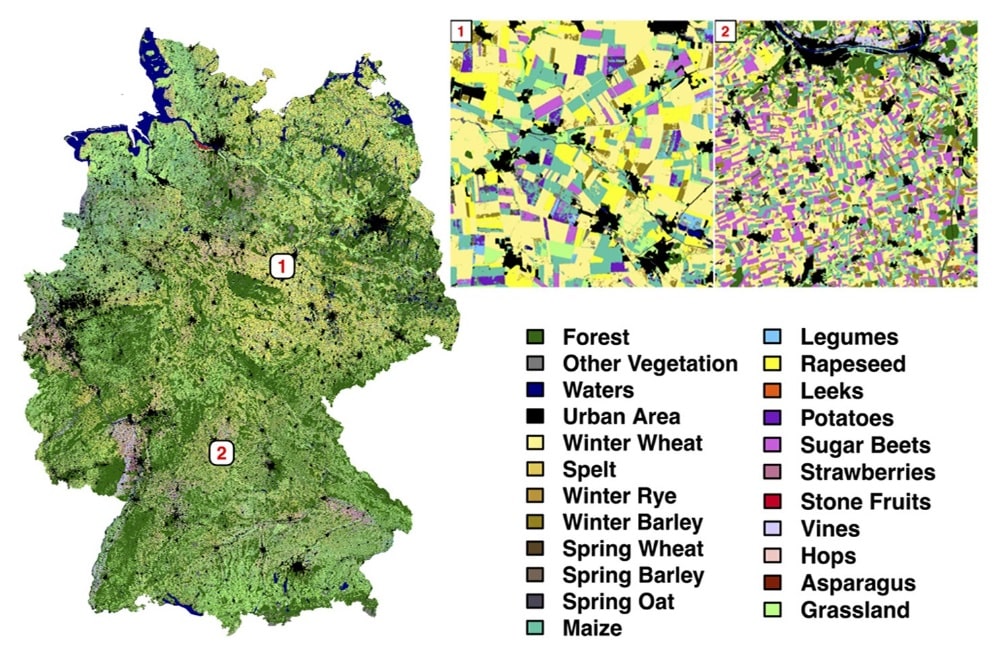
Damit die UFZ-Klassifikationsmethode lernt, Mais von Weizen oder Hopfen von Wein zu unterscheiden, stehen in der Trainingsphase vor-Ort-Informationen der Bundesländer für ausgewählte landwirtschaftliche Flächen bereit. Für einzelne Standorte ist also bekannt, welche Feldfrucht auf welchem Acker gedeiht. Für die flächendeckende Bestimmung der Landbedeckung, haben die Wissenschaftler Deutschland in sechs Regionen unterteilt.
“In der Magdeburger Börde werden andere Feldfrüchte angebaut als im Rheingau”, erklärt Preidl. “Außerdem wächst ein und dieselbe Nutzpflanzenart im Breisgau anders als in der Uckermark. Klima und Höhenlage haben einen großen Einfluss.” Das Ergebnis: Der Algorithmus der Forscher erkennt 19 verschiedene Feldfruchtarten mit einer Genauigkeit von 88 Prozent. Bei den Hauptfeldfrüchten liegt die Trefferquote sogar über 90 Prozent.
Unter Berücksichtigung von rund 7.000 Satellitenbildern, haben sie nun eine Deutschlandkarte der landwirtschaftlichen Nutzflächen zunächst für das Jahr 2016 generiert. Diese enthält neben der eigentlichen Landbedeckung auch eine Aussage über die Klassifikationsgüte, also mit welcher Genauigkeit das System die jeweilige Pflanze für ein bestimmtes Pixel erkannt hat.
Damit ist das Potenzial des UFZ-Ansatzes aber noch längst nicht erschöpft. Statt Weizen und Mais unterscheiden Preidls Algorithmen in einem Projekt mit dem Bundesamt für Naturschutz (BfN) auch Fichte, Buche und andere Baumarten voneinander. Damit untersucht er, inwieweit sich Wälder mithilfe von Satellitendaten naturschutzfachlich bewerten lassen.
“Wenn wir wissen, welche Baumarten eines Waldgebietes über die Zeit vorherrschen, lassen sich Auswirkungen von Sturmereignissen, Dürreschäden oder Schädlingsbefall besser feststellen. Ein widerstandsfähiger Wald ist im Sinne der Ziele zur nachhaltigen Entwicklung ökonomisch und ökologisch hochrelevant”, sagt Preidl.
“Unsere Methodik lässt sich aufgrund der Berücksichtigung der jeweiligen zeitlichen Abfolge von wolkenfreien Beobachtungen und Landnutzung auf andere Regionen innerhalb und außerhalb Europas und weitere Jahre übertragen”, gibt Dr. Daniel Doktor, Leiter der Arbeitsgruppe Fernerkundung im Department Landschaftsökologie des UFZ einen Ausblick auf die nächsten Schritte.
“Wenn diese Methodik mit anderen Modellen – beispielsweise zur Phänologie oder zur Ökologie – verschnitten wird, lassen sich nicht nur Aussagen bezüglich artenspezifischer Verwundbarkeit gegenüber Extremereignissen wie Dürren treffen, sondern auch zum zukünftigen Verhalten von Ökosystemen als Kohlenstoffquelle oder -senke”, so Doktor.
Publikation:
Sebastian Preidl, Maximilian Lange, Daniel Doktor, (2020) Introducing APiC for regionalised land cover mapping on the national scale using Sentinel-2A imagery, Remote Sensing of Environment, Volume 240, Article 111673, https://doi.org/10.1016/j.rse.2020.111673
Deutschlandkarte Landbedeckung: www.ufz.de/land-cover-classification
(Nicht) Demonstrieren in Corona-Zeiten






Keine Kommentare bisher